단일 세포 분석(생명과학)
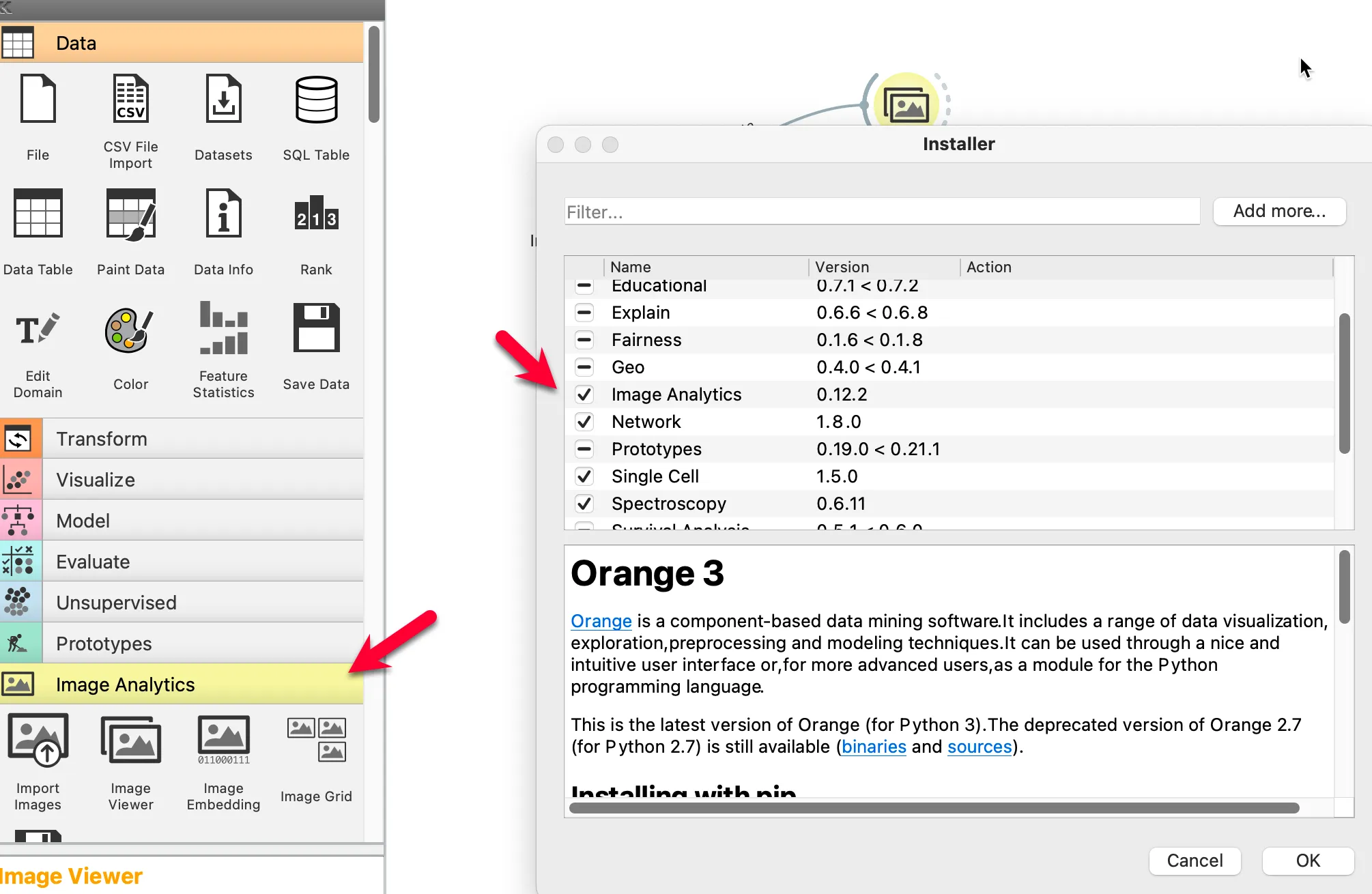
1. 실습 준비하기
두 개의 위젯이 준비되었다면 실습을 위한 준비가 완료되었습니다.
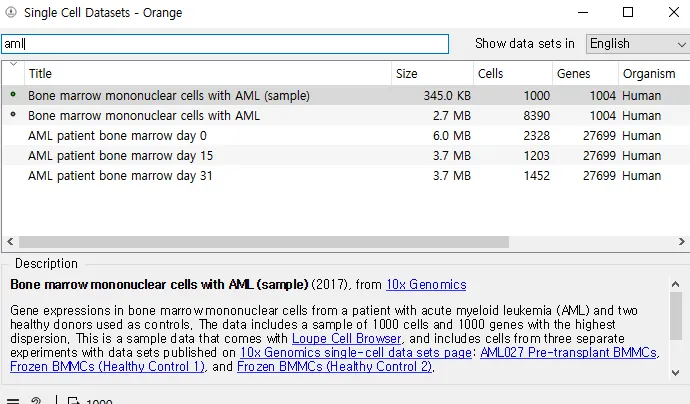
2. 단일 세포 데이터 살펴보기
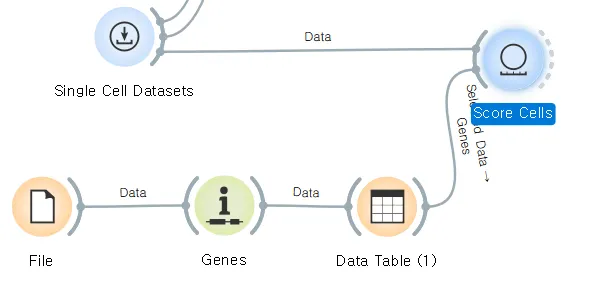
Single Cell Datasets에는 기본 위젯 중 하나인 Datasets과 동일하게 단일 세포 데이터 세트의 데이터들이 담겨있습니다.
AML(Acute myeloid leukemia)이란?
급성 골수성 백혈병이며 특정 유형의 백혈구 암입니다. AML은 뼈 안의 해면 조직인 골수에서 시작되는 병입니다.
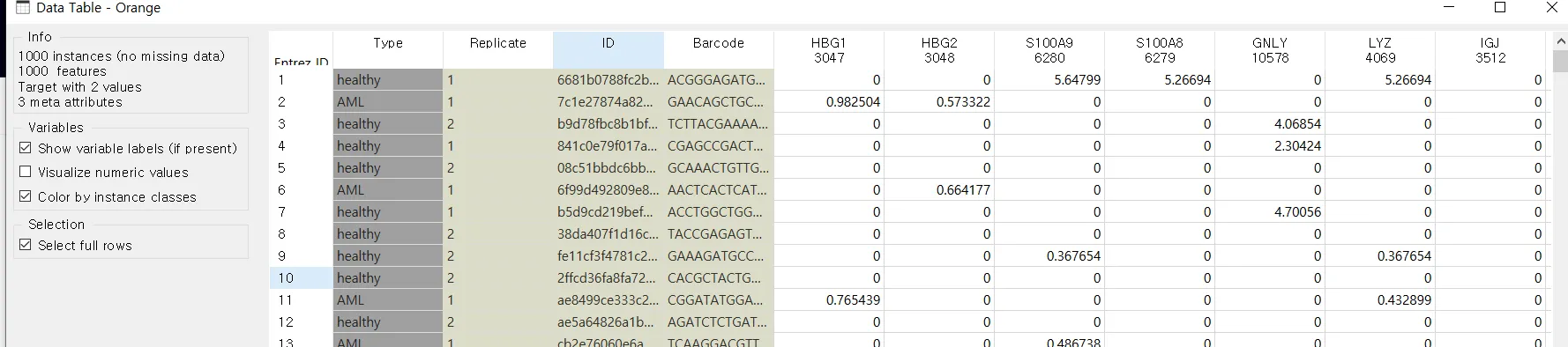
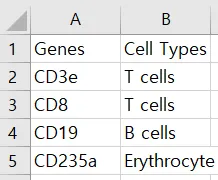
첫 번째 열 Type: 세포가 건강한 기증자인지, 백혈병 환자의 것인지를 나타내는 정보
두 번째 열 Replicate: 세포의 복제 아이디어에 대한 정보
세 번째 열 ID: 세포 ID
네 번째 열 Barcode: 세포 바코드
•
열의 나머지 부분에 저장된 유전자 표현은 필터링 되지 않은 단일 세포 데이터가 드물기 때문에 많은 0을 포함합니다.
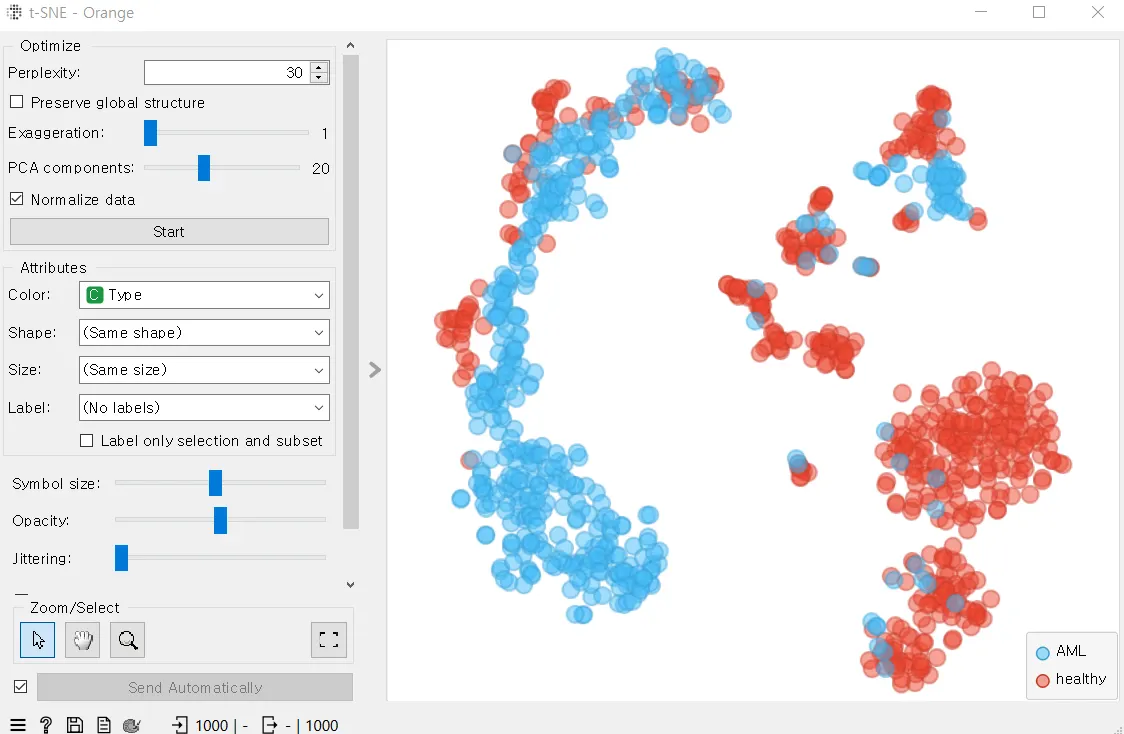
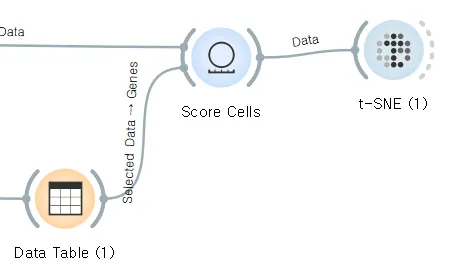
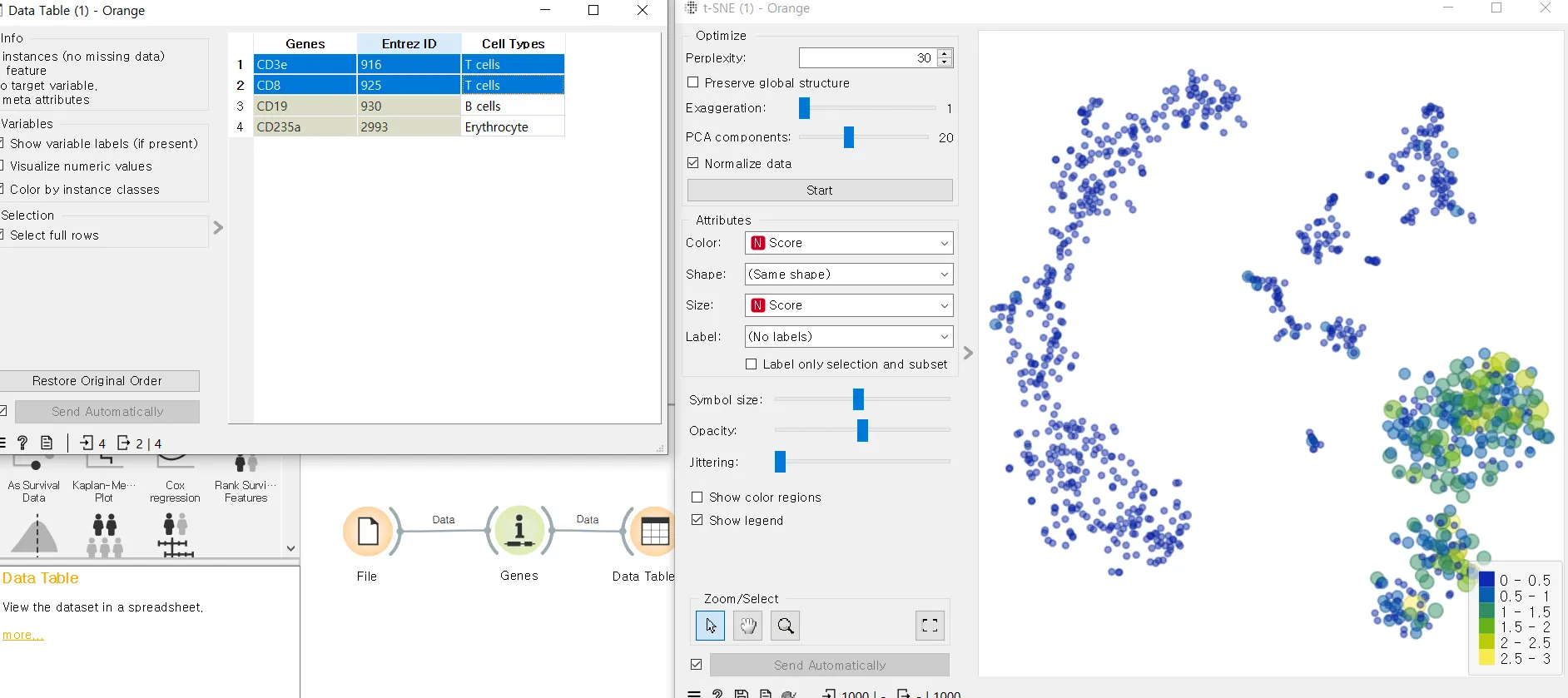
연결된 t-SNE 위젯을 클릭하면 아래 이미지가 나타납니다. AML(백혈병 기증자)과 healthy(건강한 기증자) 중 어느 기증자의 세포인지 시각화되어 구별이 잘 되어 있는 것을 확인할 수 있습니다.
t-SNE(t-Stochastic Neighbor Embedding)이란?
높은 차원의 복잡한 데이터를 2차원에 차원 축소하는 방법이다.
3. 단일 세포 데이터 분류해 보기
제공되는 Type에 따라 구별되는 것을 넘어 유전자에 따라 세포 유형으로 구별해 보려고 합니다.
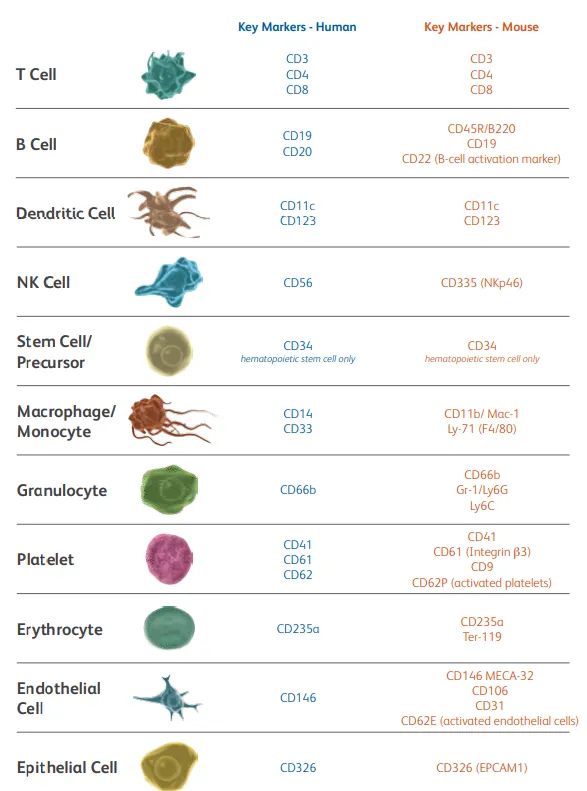
⇒ 2페이지를 보면 이미지처럼 유전자에 따라 세포 유형을 확인할 수 있습니다.
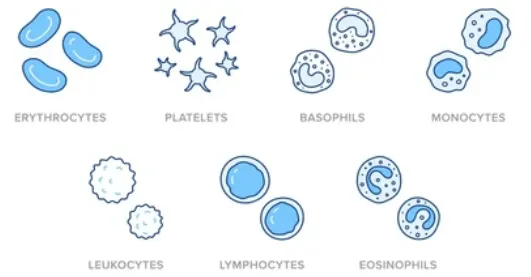
ex) Erythrocyte(적혈구) - CD235a
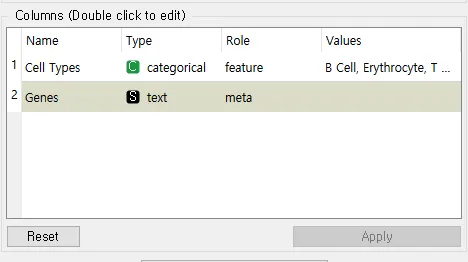
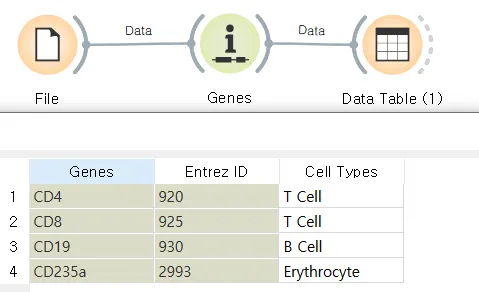
BD CD Marker Handbook에 있는 Key Markers - Human 중 일부를 가져와서 표를 작성합니다.
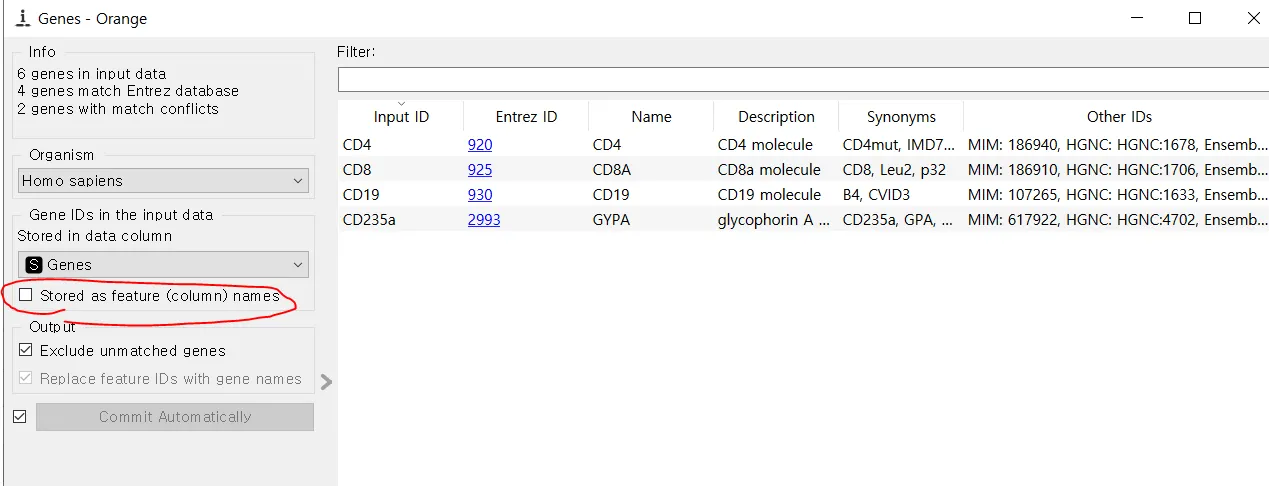
Genes를 더블 클릭하고 Stored as feature (column) names 열 이름으로 저장하지 않도록 체크를 해제합니다. NCBI 데이터베이스에서 발견되어 입력한 ID를 Entrez ID로 변환합니다.
Entrez란?
NCBI에서 운영하고 있는 서열 데이터베이스입니다.
※ NCBI: 국립생명공학정보센터로 국립보건원(NIH) 산하 국립의학도서관(NLM)의 한 부서입니다. NCBI는 방대한 생물학적 데이터베이스와 자원 모음을 조직하고 유지하여 과학계가 무료로 사용할 수 있도록 하는 역할을 합니다.
•
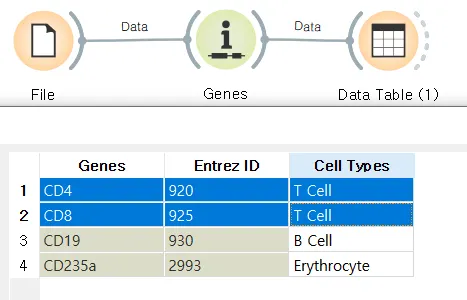
Data Table에서 T 세포를 누른 경우
•
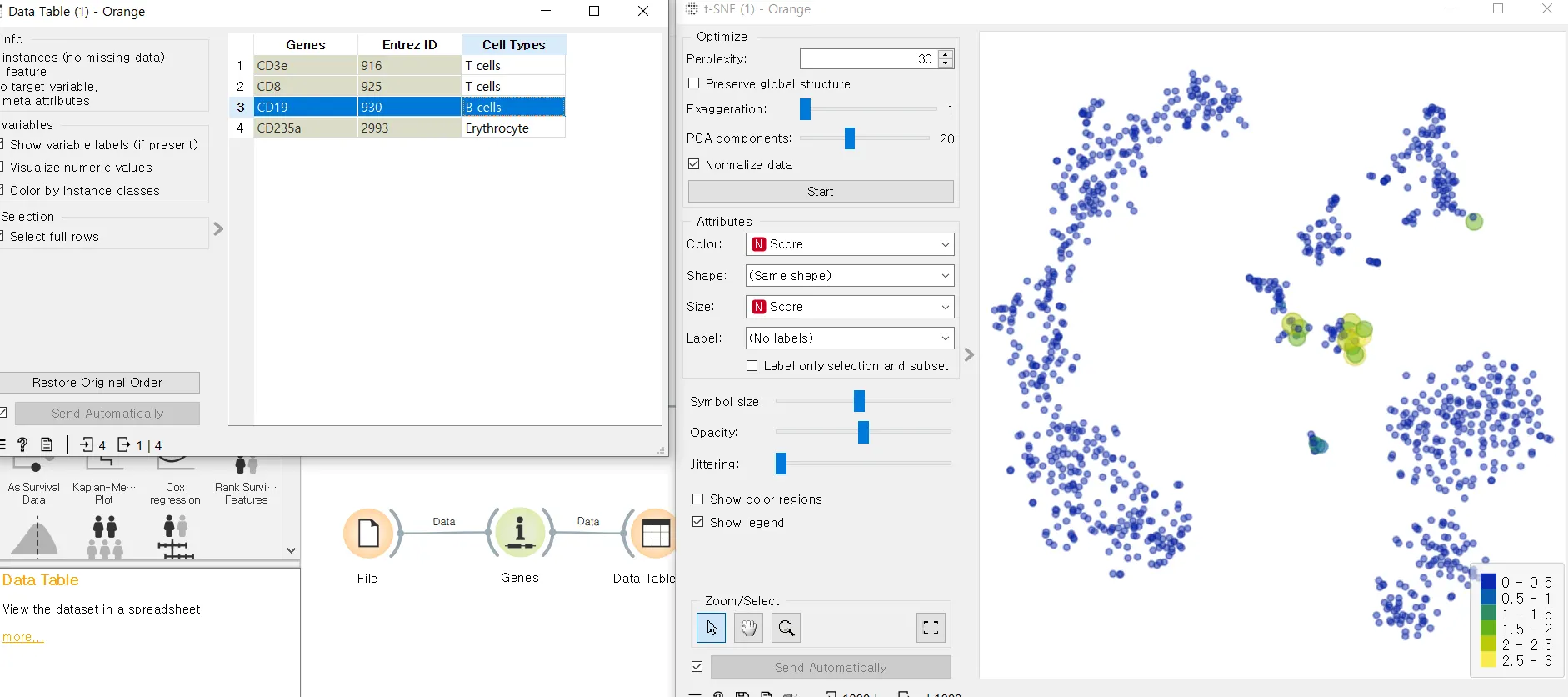
Data Table에서 B 세포를 누른 경우
•
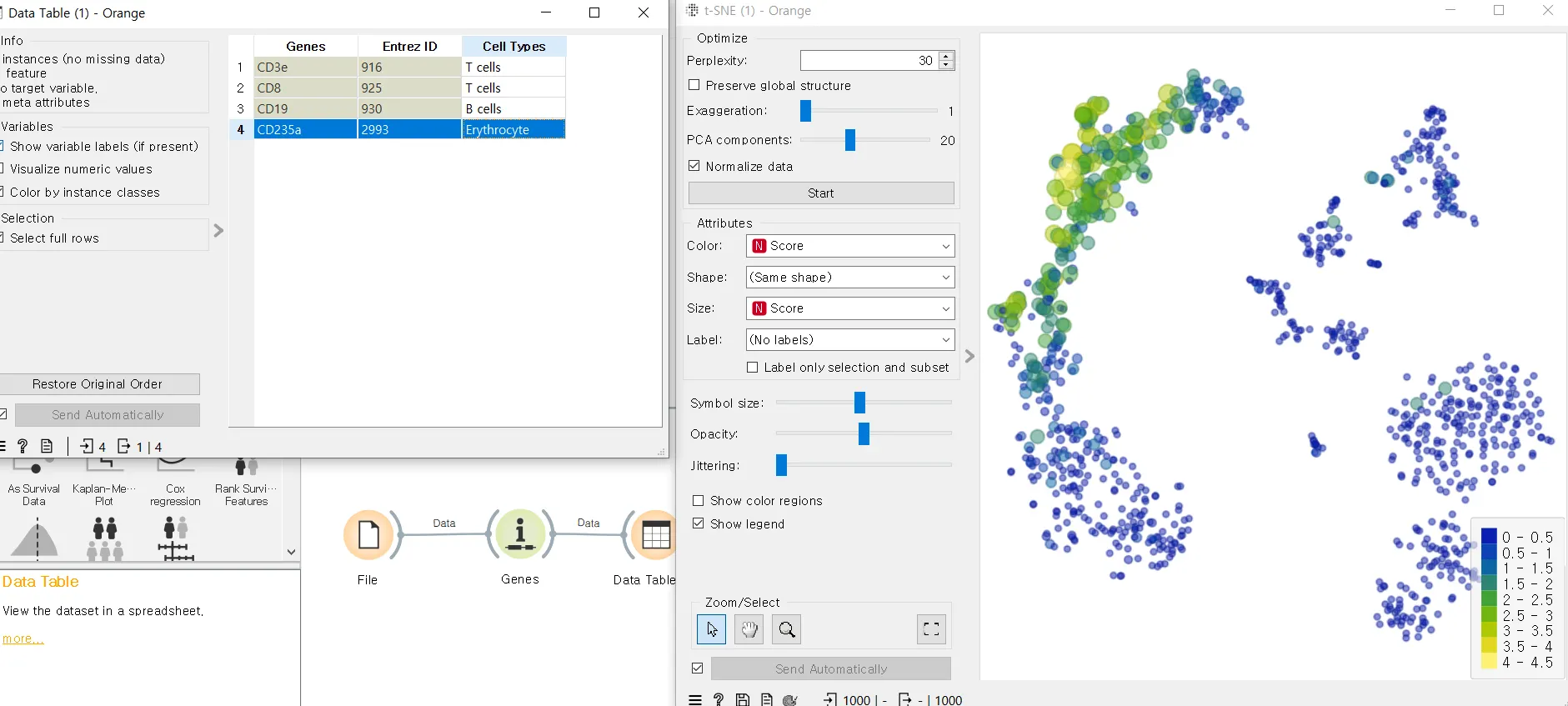
Data Table에서 적혈구를 누른 경우
이렇게 같은 세포끼리 클러스터링이 된 것을 알 수 있습니다.
오렌지3에서 지원하는 Bioinformatics(생물정보학)와 Single Cell(단일 세포)에 대해서 알아보았습니다. 이 위젯들은 생명과학 교과목과 융합 수업하거나 동아리 시간에 활용하기 좋을 것 같습니다. 데이터를 분석하고 시각화해 보면서 학생들과 재미있는 수업하셨으면 좋겠습니다 감사합니다
감사합니다
구름 이미지 분석(지구과학)
1. 문제 확인하기
상황
구름은 지상에서 증발한 수증기로 이루어져 있고, 때로는 대기 상층의 빙정과의 혼합물이기에 구름의 종류는 단기 기상 예측에 중요한 지표 중 하나이다. 구름의 종류를 분류하는 방법 중 하나는 위성 영상을 이용하는 것인데, 이 경우 가시광선 영상, 적외선 영상 등 여러 이미지를 동시에 육안으로 구분해야 하며, 일상 생활에는 적용하기 어려운 점이 있다. 구름 이미지 사진을 이용하여 구름의 종류별로 쉽게 분류할 수는 없을까?
정의
지상에서 촬영한 구름 이미지를 트리 기반 분류 모델을 이용하여 종류별로 구분해 보자.
배경 지식
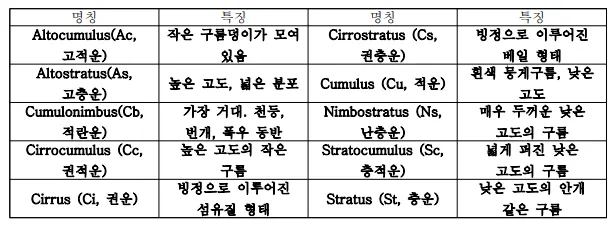
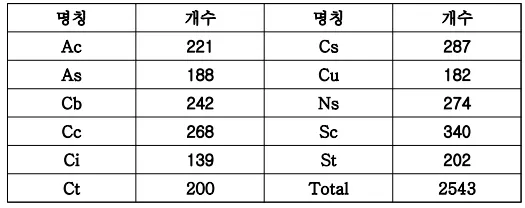
구름은 생성 과정과 시간에 영향을 받아 그 형태가 달라지고, 다른 종류의 구름으로 생성된다. 구름의 종류는 총 10개로, 아래의 표와 같다.
2. 데이터셋 준비하기
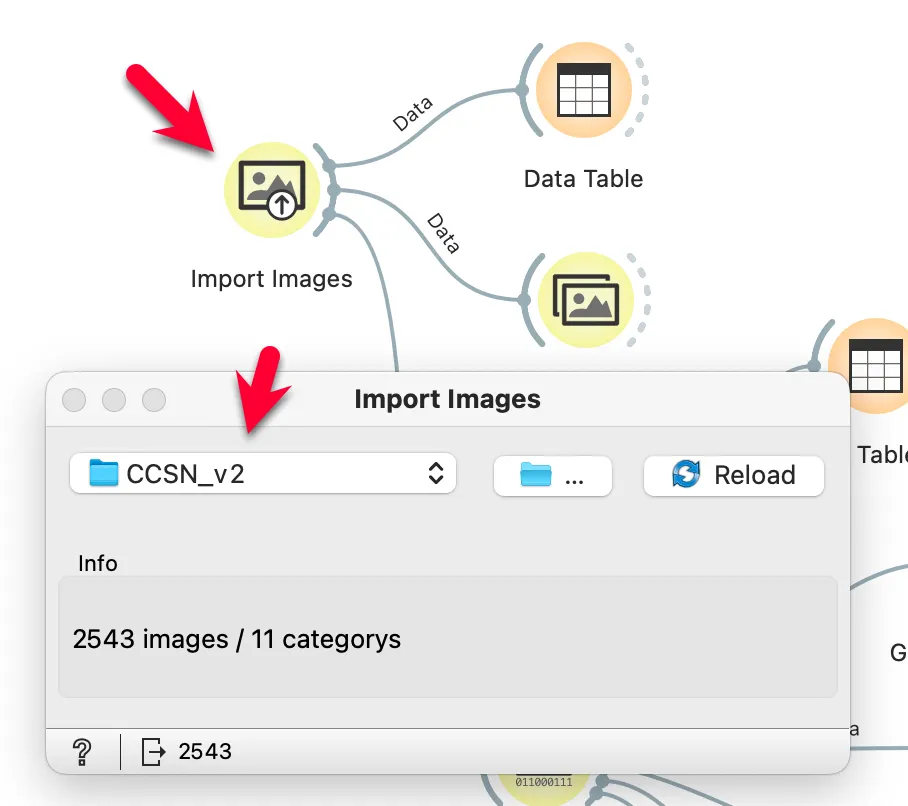
지상 구름 이미지 데이터 세트인 CCSN(Cirrus Cumulus Stratus Nimbus) dataset을 이용합니다.
CCSN dataset은 총 2543개의 400x400 픽셀의 구름 이미지를 포함하고 있고, 구름 종류별 데이터 개수는 아래의 표와 같습니다.
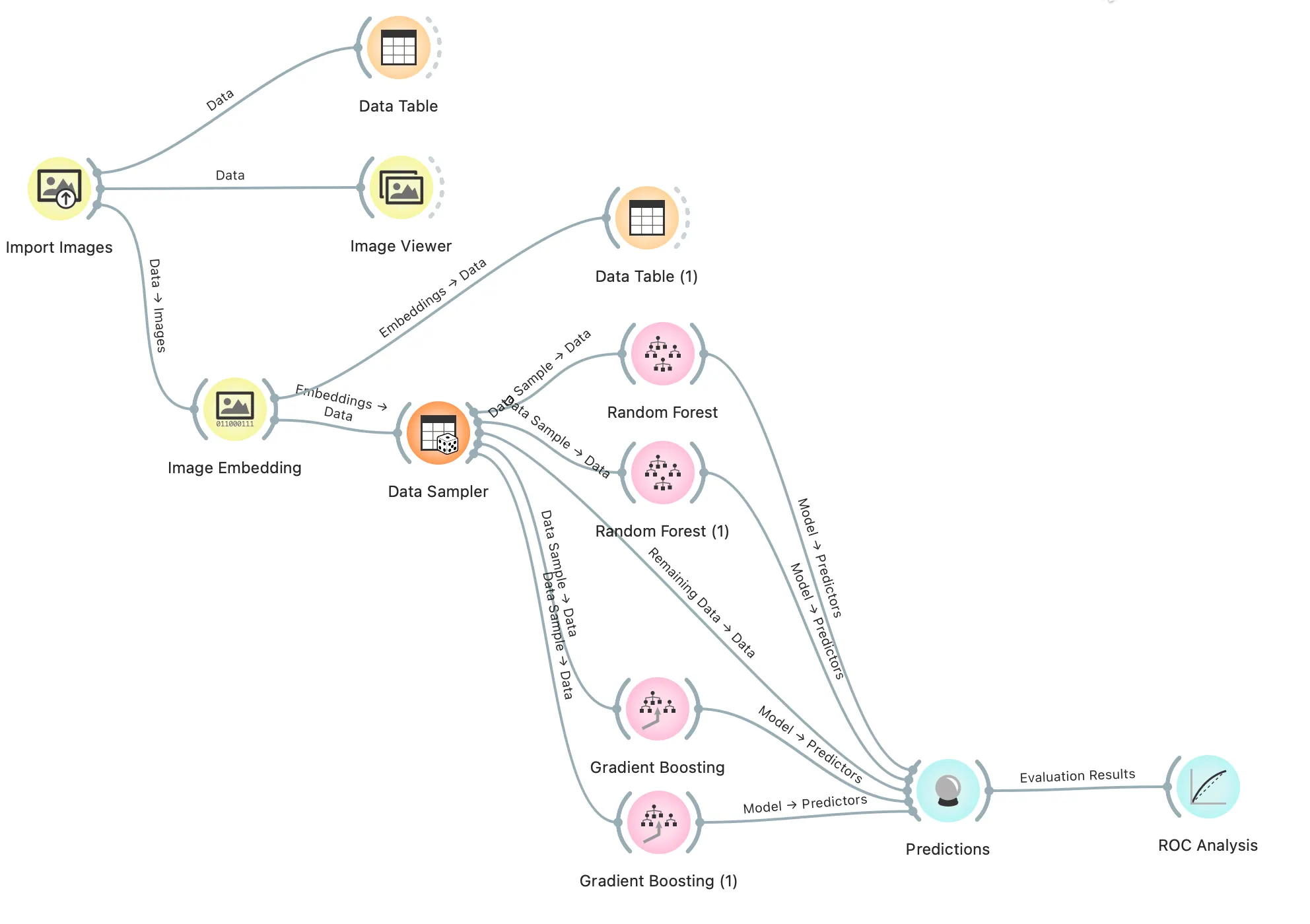
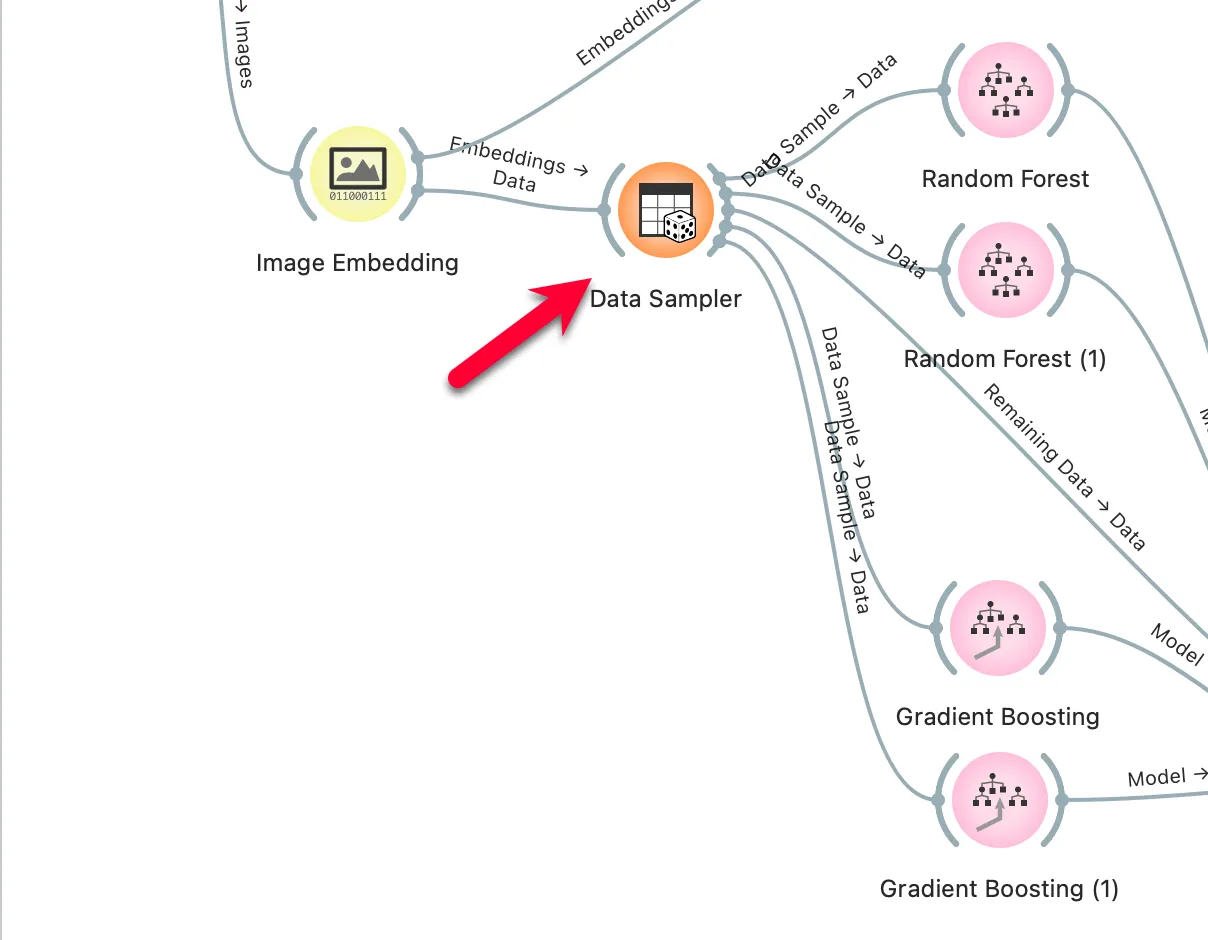
전체 위젯 구성
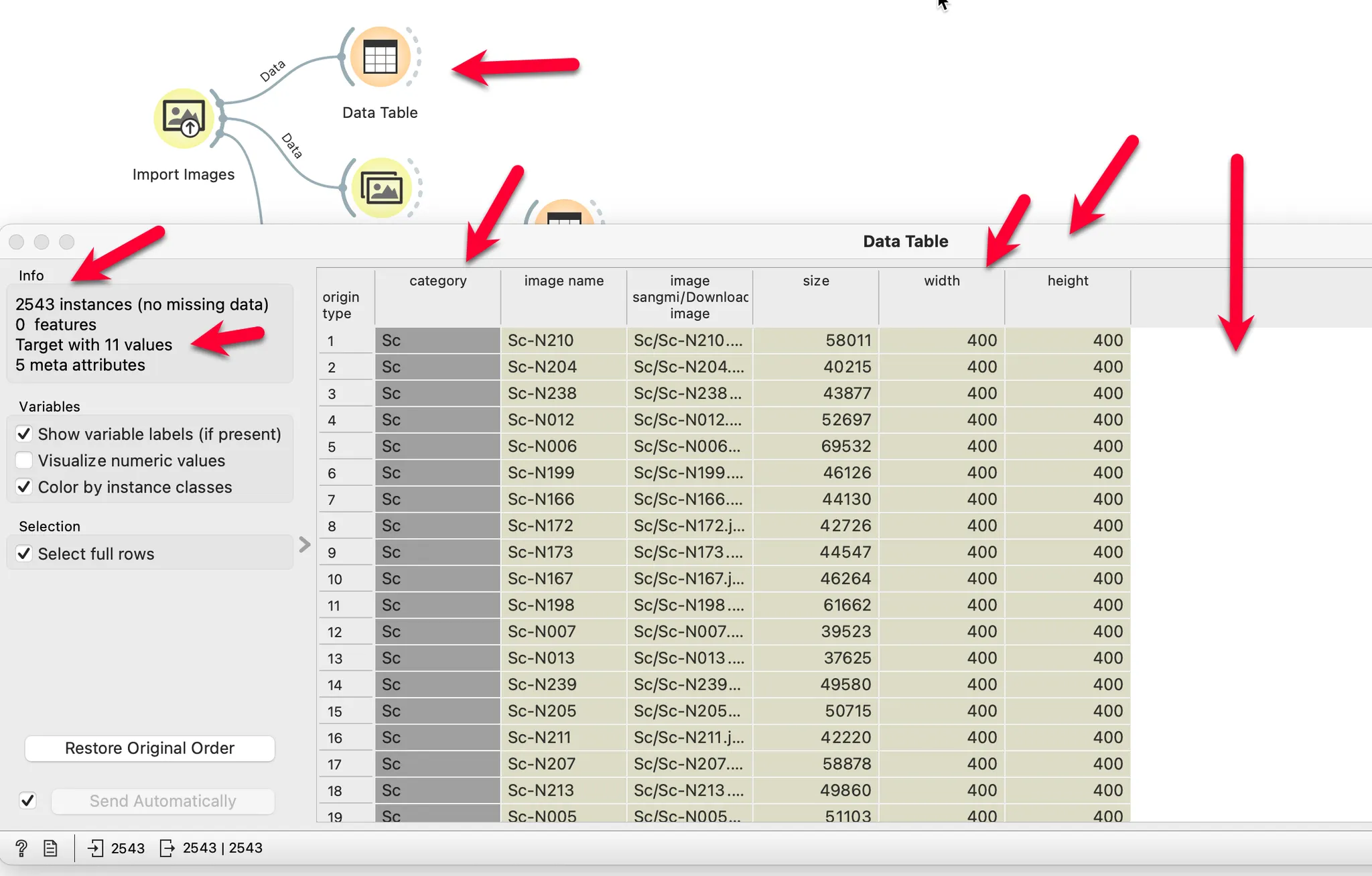
3. 데이터 불러오기
# 데이터셋의 정보를 알 수 있다. 2543개의 데이터를 가지고 있으며 타겟은 11개이다.
Markdown
복사
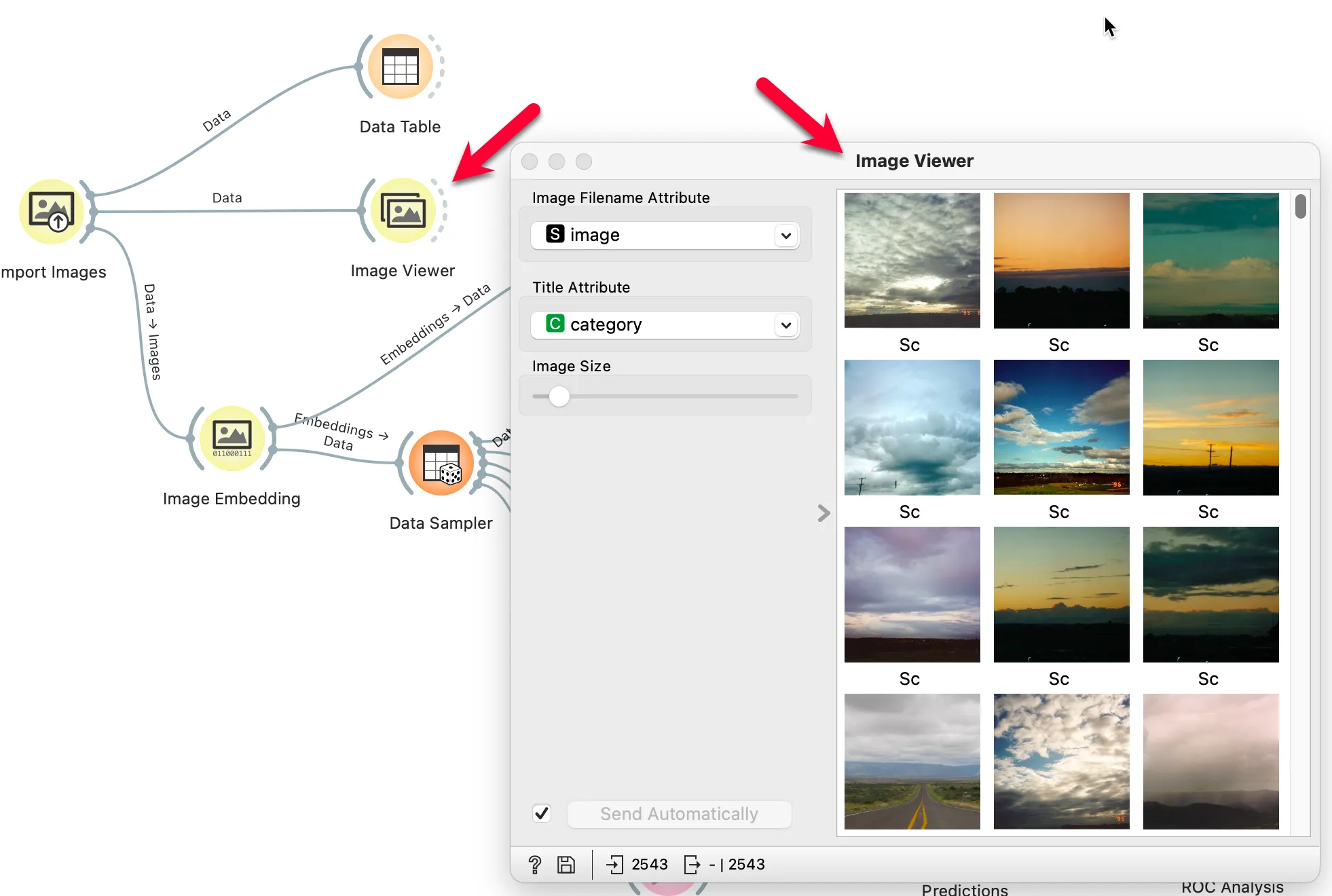
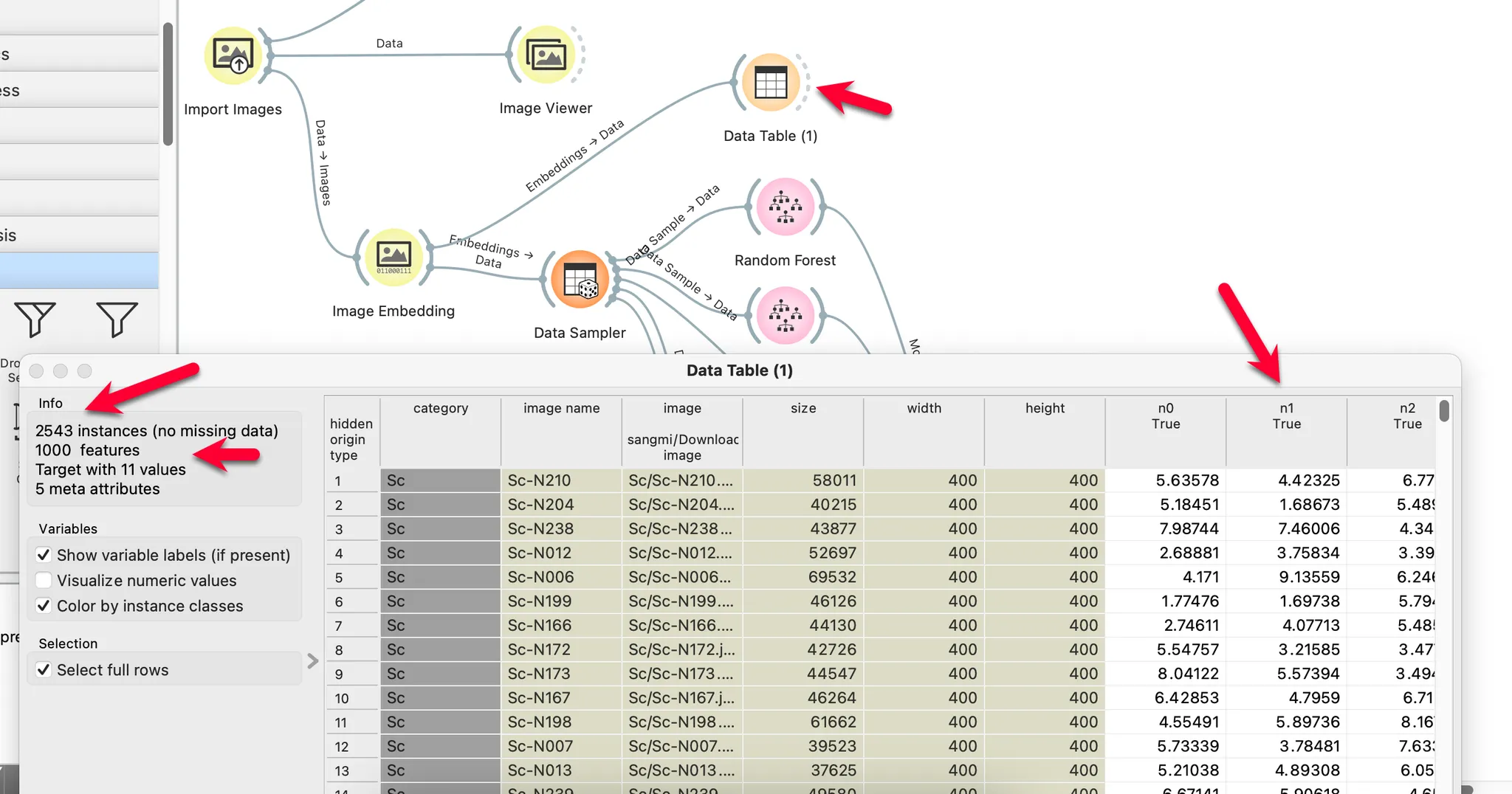
4. 이미지 분석하기
# Data Table 위젯에서는 특성(Features)이 0개 였지만 Data Table(1) 위젯에서는 1000개의 특성이 있다.
# 데이터 테이블을 오른쪽으로 스크롤을 해보면 n0 ~ n999까지의 특성이 생성된 것을 알 수 있다.
# 이러한 특성은 이미지 파일의 화소를 1000개의 특성으로 나눠서 각 특성에 숫자 값을 준 것이다.
Markdown
복사
5. 트리 기반 분류 모델 학습하기
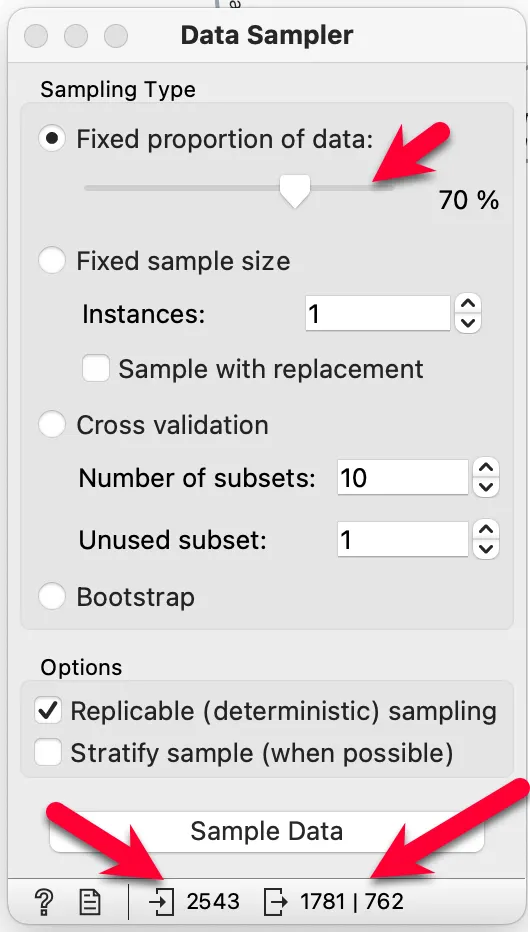
기본 값이 70%로 되어 있습니다. 훈련 데이터를 데이터셋의 70%, 나머지를 테스트 데이터로 사용할 수 있도록 데이터를 구분합니다.
트리 기반 분류 모델
결정 트리 모델을 기반으로 파생된 모든 모델을 포함한다. 머신러닝에서는 여러 모델의 결과를 종합해서 더 좋은 성능을 내는 방법을 앙상블(ensemble) 기법이라고 한다. 앙상블은 보팅(voting), 배깅(bagging), 부스팅(boosting)방식이 있다. 보팅과 배깅은 기본 모델을 여러 번 수행하여 평균이나 다수결 투표로 결과를 내는 방식이다.
랜덤 포레스트
다수의 결정 트리 모델을 생성해서 다수결 혹은 평균을 적용하여 조합한 모델이다. 동일한 학습 데이터세트에서 각기 다른 입력 변수(feature)와 각기 다른 데이터 샘플을 사용하여 여러 개의 소규모 트리들을 생성한 후 조합한다. 다수의 결정 트리 모델의 조합을 통해 과적합을 방지하지만 결과의 해석이 어려운 면이 있다.
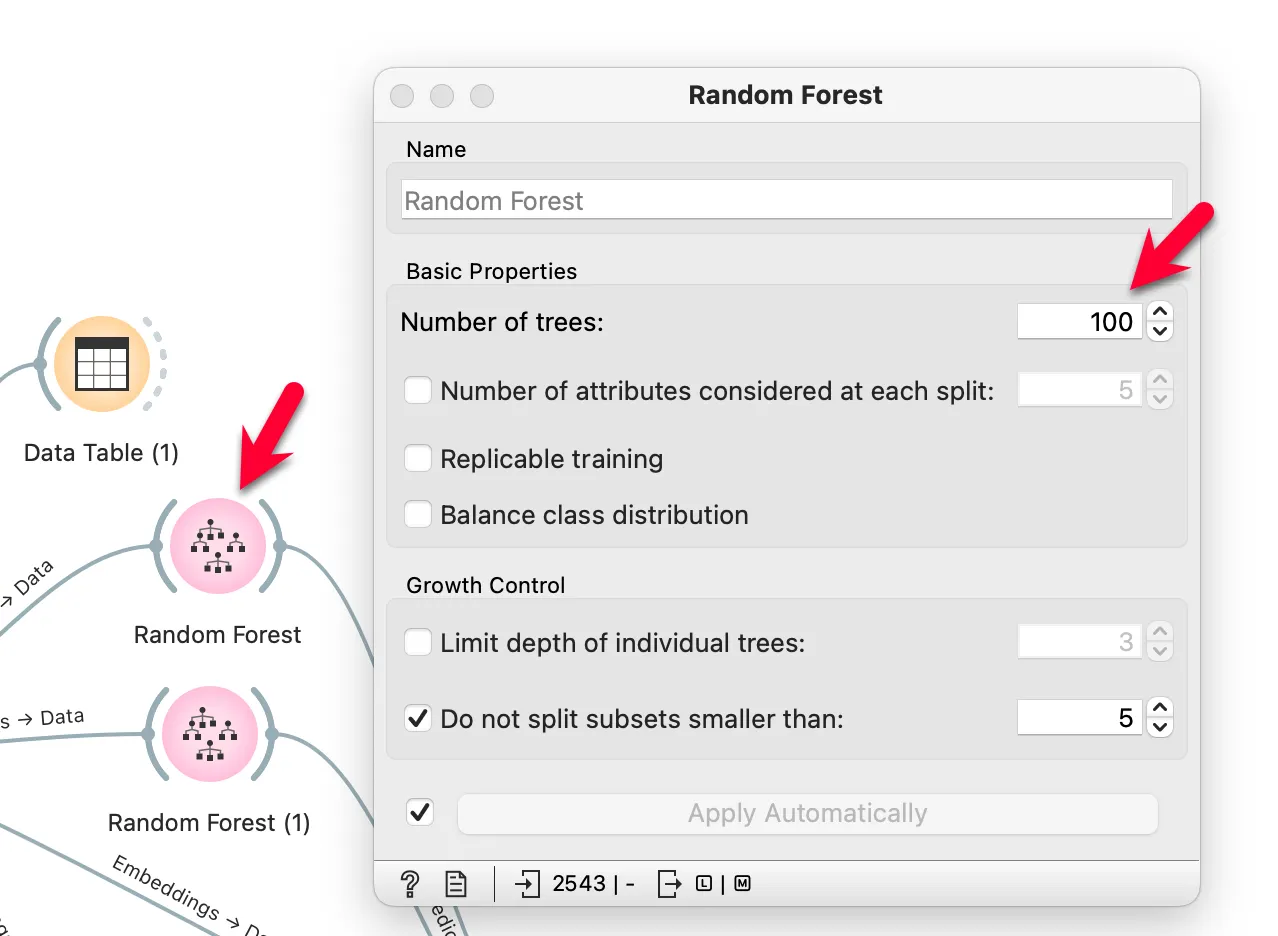
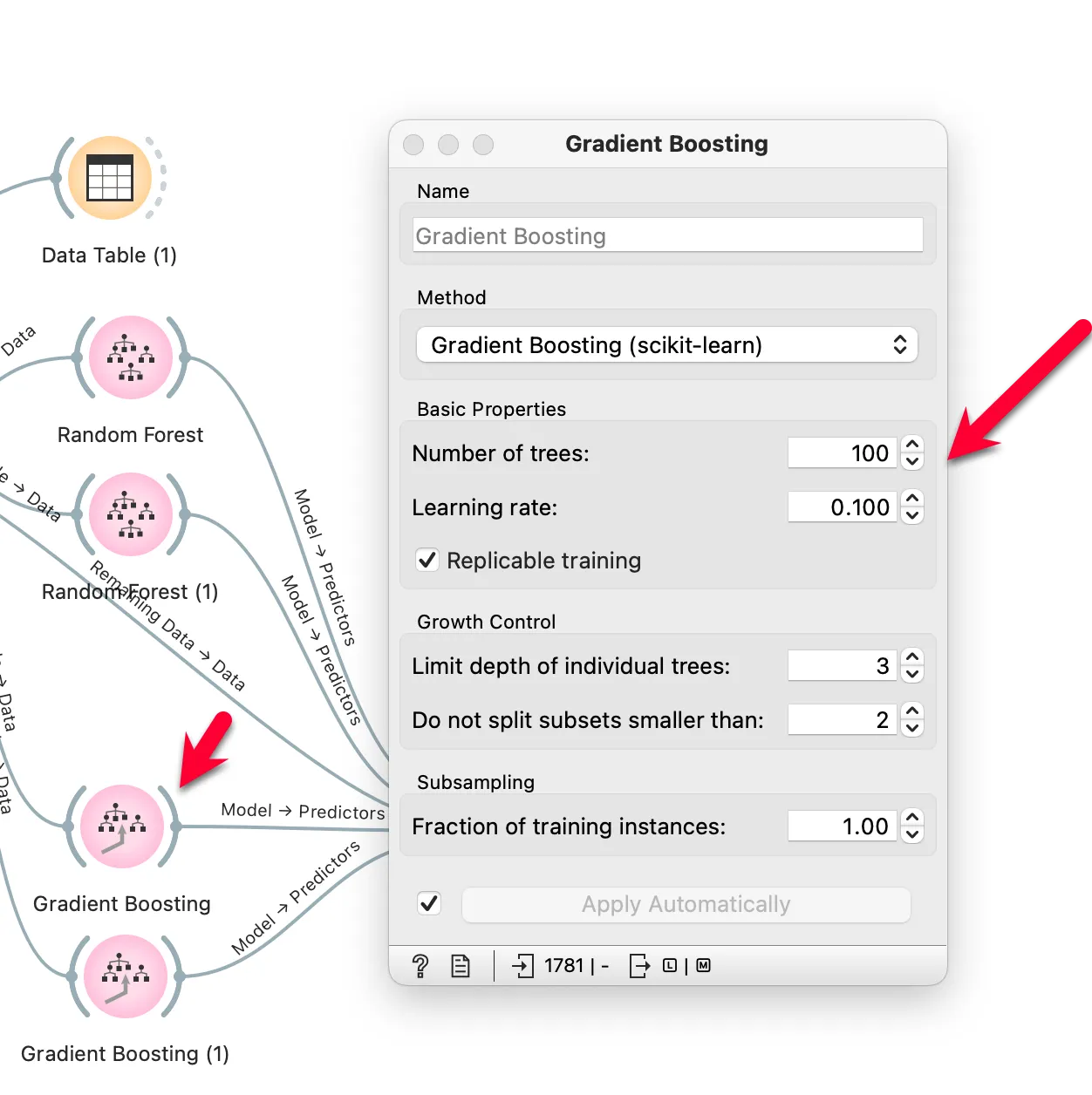
첫 번째 [Model - Random Forest] 위젯을 더블 클릭하여 Number of trees의 값을 100으로 변경합니다.
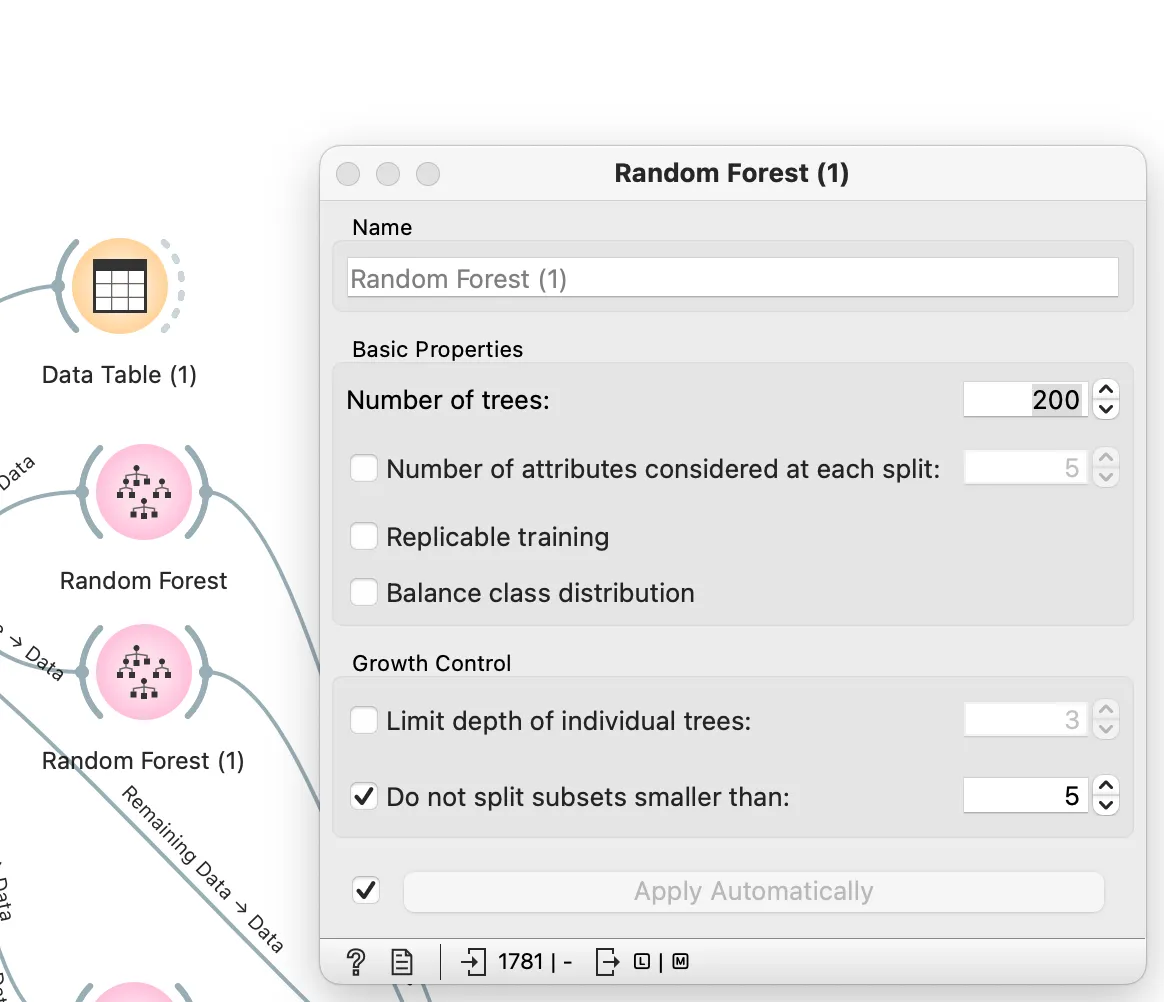
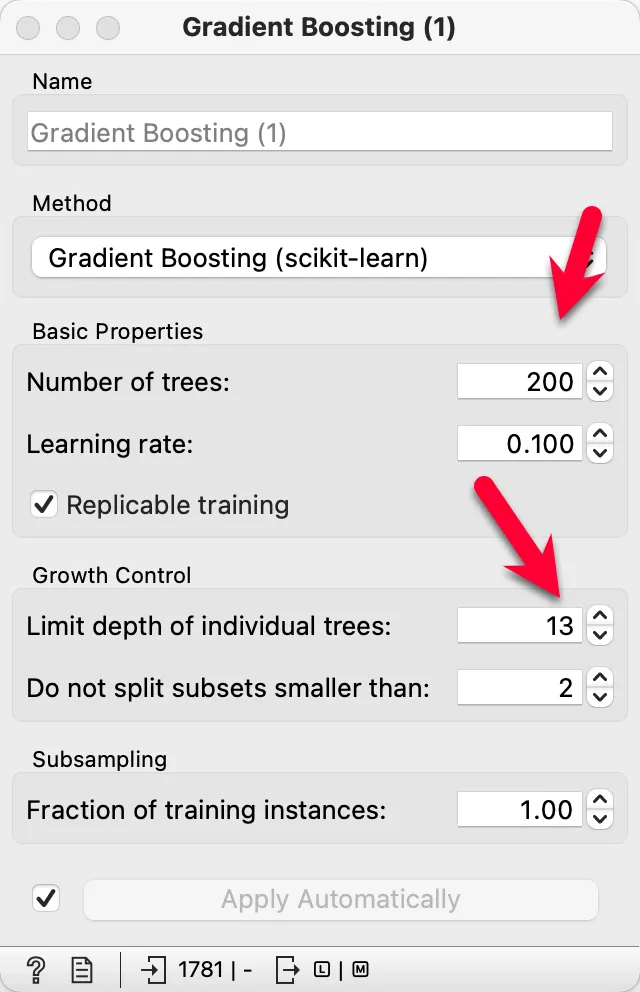
두 번째 모델은 200으로 값을 바꾸고, 파라미터 값으로 넣은 값을 임의로 바꾸어 가면 찾아봅니다.
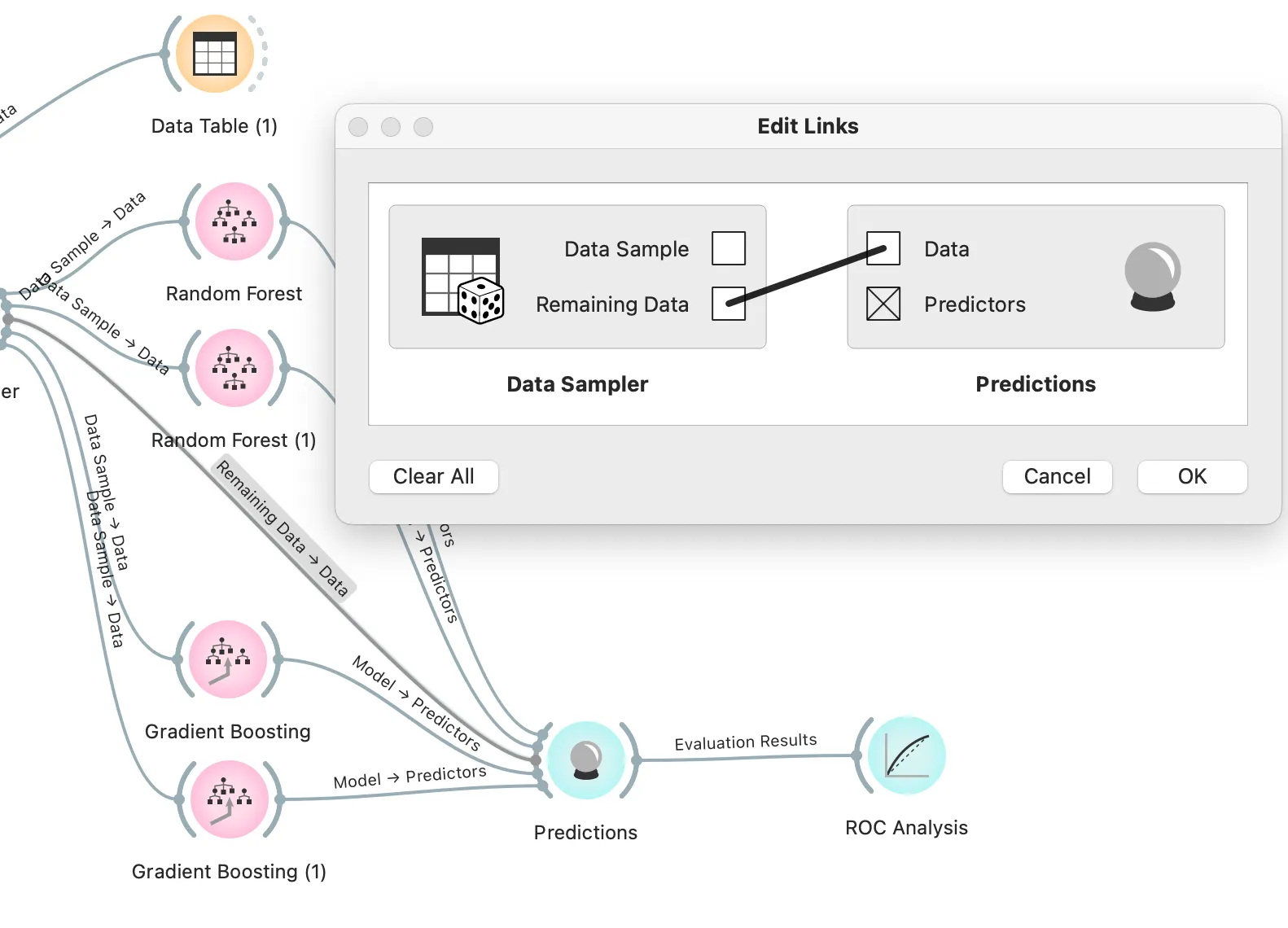
6. 평가 결과 분석
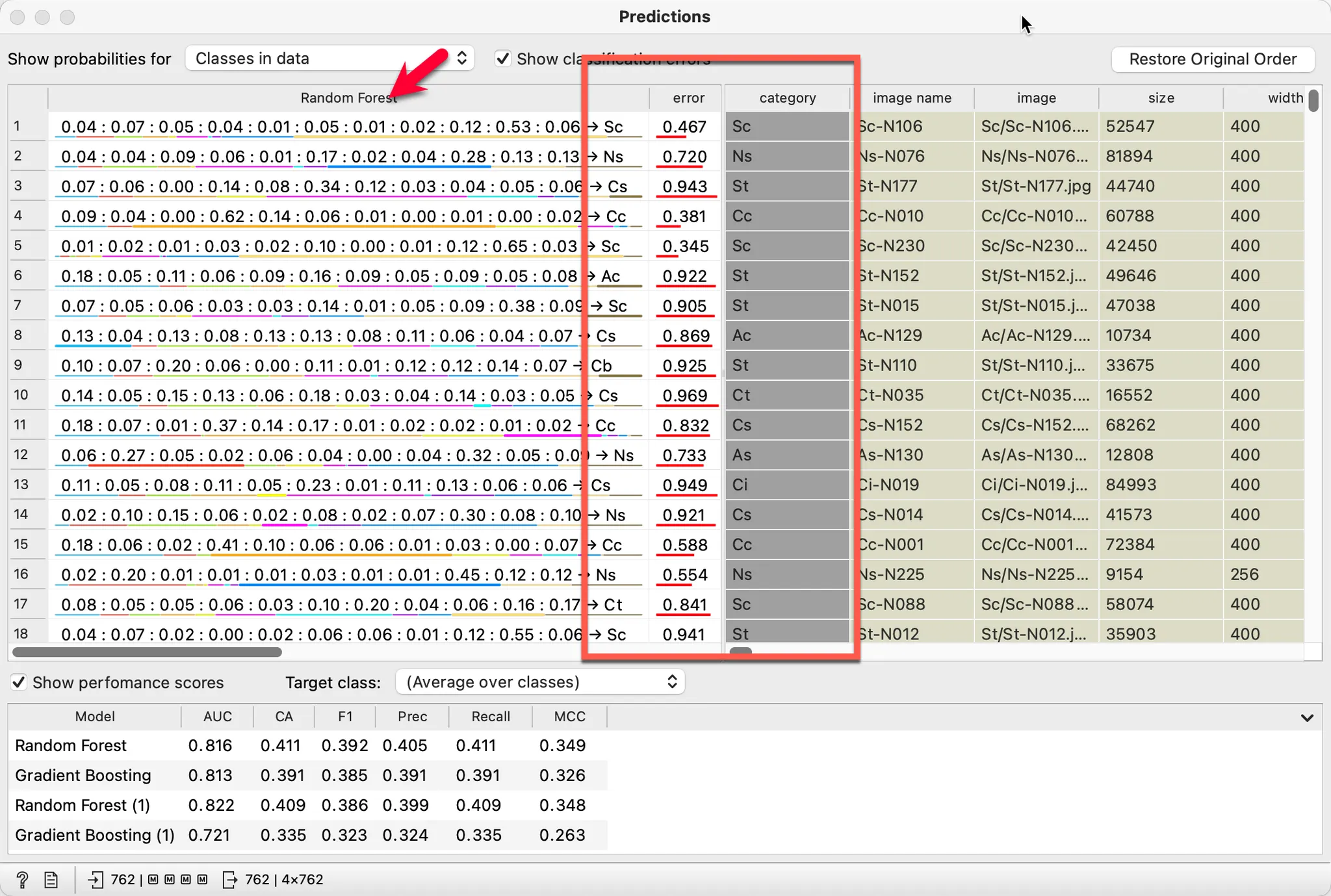
# Predictions의 실행 결과는 매번 다를 수 있다.
# 오렌지3는 머신러닝 모델에 random_state를 지정하는 기능이 없으므로 앞으로 실행하는 모든 결과는 다르게 나올 수 있다.
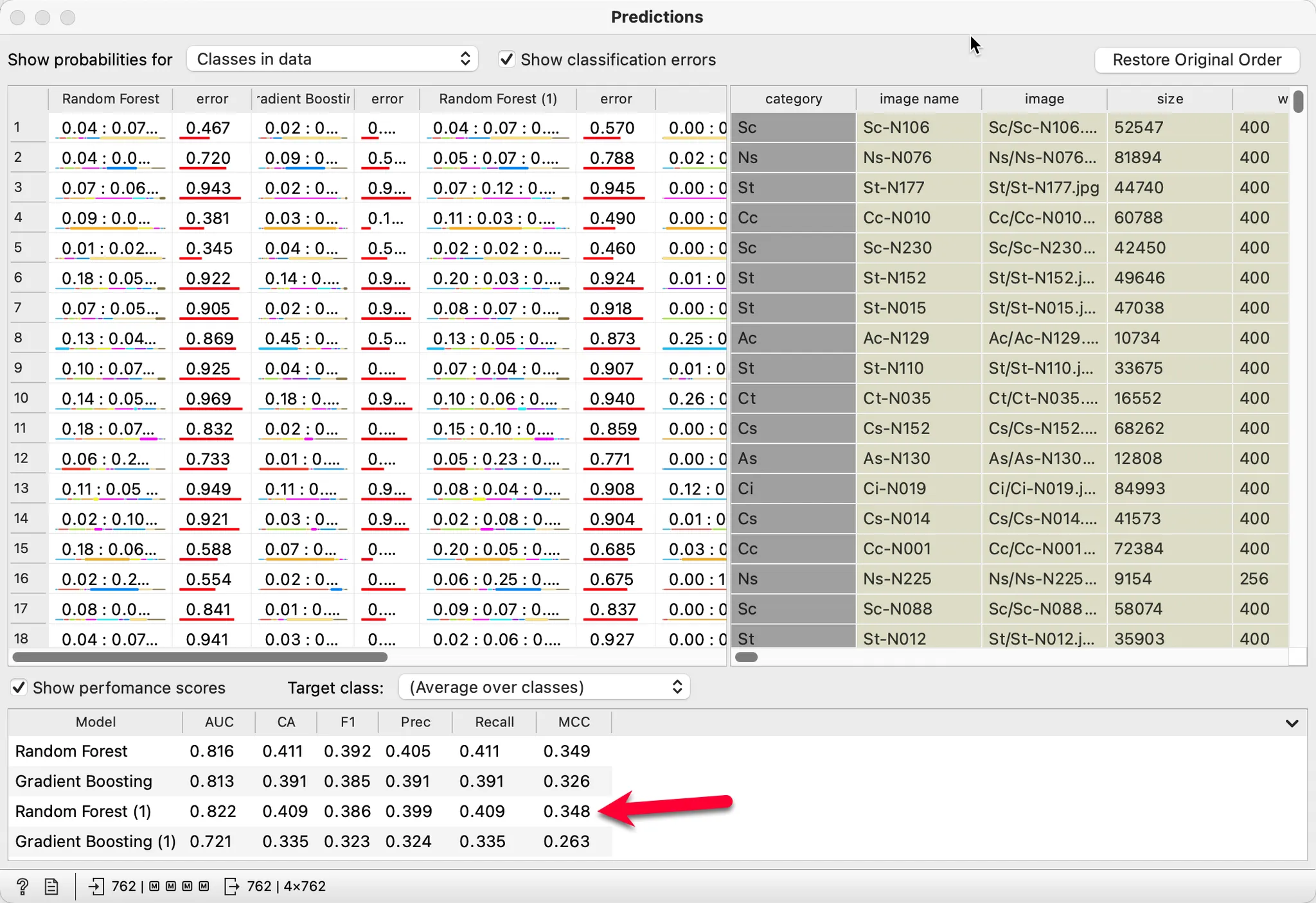
# AUC가 1에 가까울 수록 좋은 모델이다.
Markdown
복사
# 4개의 모델에 대해 데이터 별로 예측한 결과를 확인 할 수 있다.
# 그 중 위의 그림에서 첫번째로 보이는 Random forest를 살펴보면 세번째 그림이 정답을 St이나 Cs로 잘못 예측한 것을 알 수 있다.
Markdown
복사
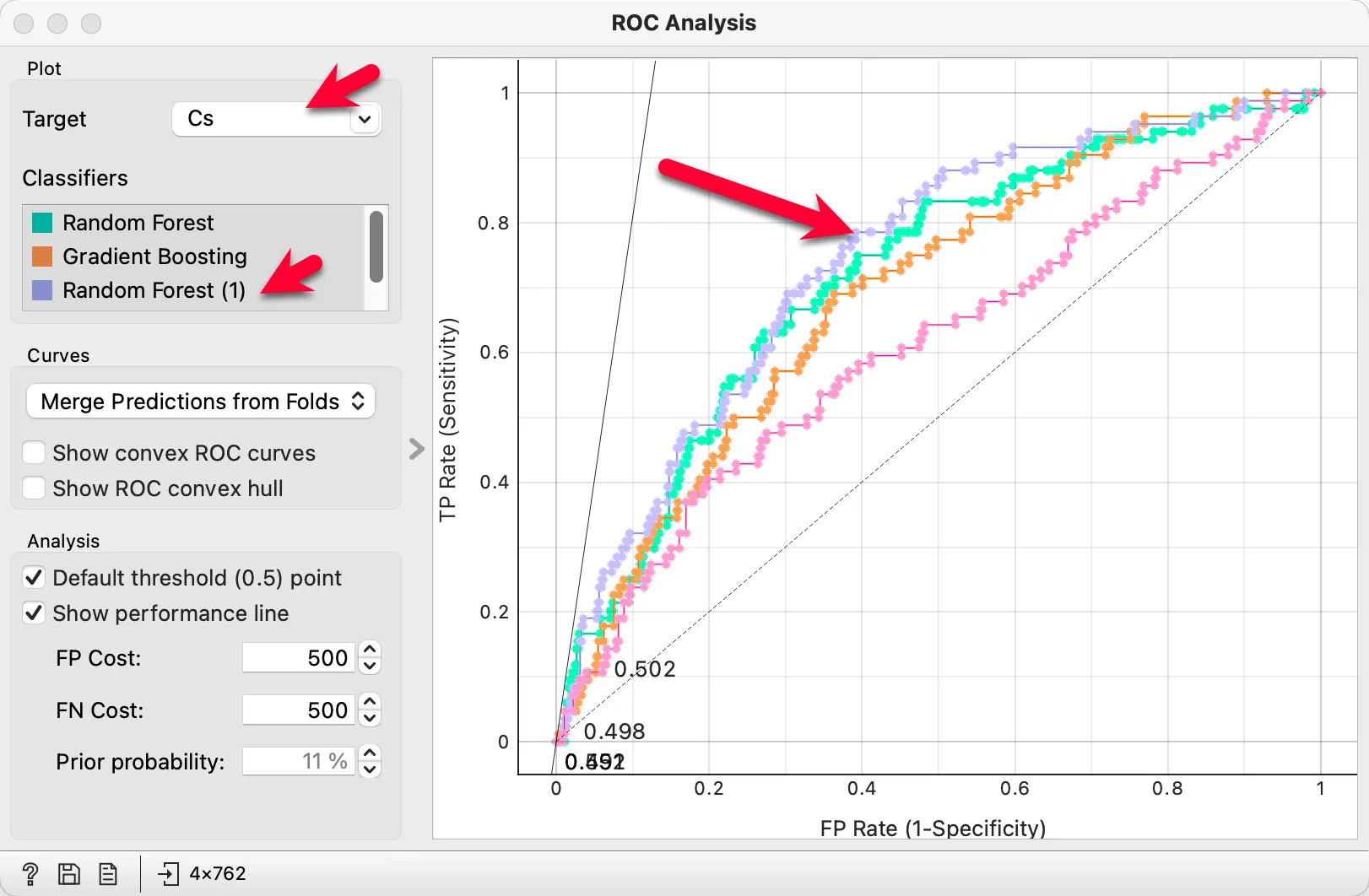
# ROC 역시 1에 가까울 수록 좋은 모델이다.
Markdown
복사
이 과정을 거치면 이미지를 분류 모델로 분류할 수 있습니다.